 Score(计算结构的能量)
Score(计算结构的能量)
# Rosetta 功能——score(计算结构的能量)
# score 功能简介
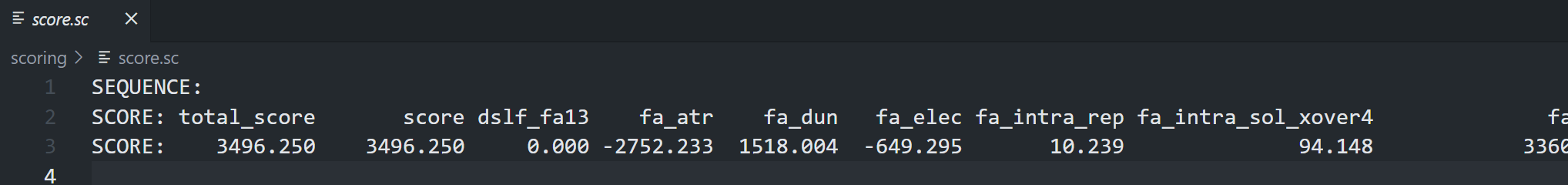
在 Rosetta 中,生物分子的能量是通过对其进行评分来计算的。 Rosetta 中的分数函数是能量项的加权和,其中一些表示物理力,如静电和范德华相互作用,而另一些则表示统计项,如在拉钱德兰空间中找到扭力角的概率。
[!info]
- fa_atr:不同残基间原子之间的 Lennard-Jones 吸引能(范德华吸引作用)。
- fa_rep:不同残基间原子之间的 Lennard-Jones 排斥能(范德华排斥作用)。
- fa_sol:Lazaridis-Karplus 溶剂化能(经验性隐式溶剂模型)。
- fa_intra_sol_xover4:同一残基内的 Lazaridis-Karplus 溶剂化能(特殊处理交叉距离大于 4 个键的原子对)。
- lk_ball_wtd:非对称溶剂化能(考虑溶剂球方向性影响的加权版本)。
- fa_intra_rep:同一残基内原子之间的 Lennard-Jones 排斥能。
- fa_elec:库仑静电势能,使用距离依赖型介电常数。
- pro_close:脯氨酸环闭合能以及前一个残基 ψ 角的能量项。
- hbond_sr_bb:一级结构中接近的主链-主链氢键能量。
- hbond_lr_bb:一级结构中距离较远的主链-主链氢键能量。
- hbond_bb_sc:主链与侧链之间的氢键能量。
- hbond_sc:侧链与侧链之间的氢键能量。
- dslf_fa13:二硫键几何约束能(用于精细建模二硫键结构)。
- rama_prepro:Ramachandran 构象偏好能(考虑前位为脯氨酸的特殊查表数据)。
- omega:主链 ω 二面角能量,用于限制肽键的共面性(约 6 度标准差的谐波约束)。
- p_aa_pp:在给定 φ/ψ 构象下某氨基酸的概率能(用于构象-序列匹配)。
- fa_dun:侧链构象能,来自 Dunbrack 统计库的旋转异构体内能。
- yhh_planarity:酪氨酸羟基保持在芳香环平面内的特殊扭转能约束。
- ref:每种氨基酸的参考能量,用于平衡氨基酸本身的内部能量(设计时尤为重要)。
- METHOD_WEIGHTS:不是能量项本身,而是
ref项所依赖的每种氨基酸的参数集。
# score 功能本地测试
输入一个 PDB 文件,输出一个打分结果。
上次更新: 2025/07/04, 10:53:42
