Rosetta介绍
Rosetta介绍
- Rosetta 平台的部署和学习,目前在本地是 docker 版本部署的,在服务器上的安装还在尝试。现在主要还在根据教程学习阶段。
- RosettaFold(一个给定序列,能够预测结构的模型,目前并不在 Rosetta 平台中)的环境部署和数据准备,目前环境基本准备好了,数据还在准备,比较大,1000G的数据。
# Rosetta 介绍
Rosetta 是一个全面的用于构建大分子结构的软件套件。作为一个灵活的多用途应用程序,它包含用于蛋白质和核酸的结构预测、设计和重建的多种工具。由 David Baker 教授领导的实验室开发,起源于1998年,是结构生物学、计算蛋白质设计和分子建模领域的核心工具之一。
# Rosetta 平台直观展示
 Rosetta每一个功能都是一个二进制文件,并不是以界面形式展示的是以脚本代码驱动的。
Rosetta每一个功能都是一个二进制文件,并不是以界面形式展示的是以脚本代码驱动的。
# Rosetta 教程
# Home
https://docs.rosettacommons.org/docs/latest/Home
# Tutorials

# Rosseta 基础规定
# Rosseta 输入
输入文件格式:
- PDB 文件(ATOM、HETATM、TER 记录,忽略部分原子编号和元素符号,默认加载残基的第一个构象)、
- PDB 列表(使用
in:file:l选项)、 - Silent 文件(Rosetta 特定格式,使用
in:file:silent选项)。
[!note] Silent 文件是 Rosetta 特有的一种紧凑格式文件,用于存储多个结构的信息。它在进行大量结构输出的模拟时特别有用,因为在这种情况下,许多文件系统在执行批量操作时会遇到问题。
# Rosseta 输出
常见结构输出文件:
- PDB 文件:Rosetta 的默认输出格式,对于不默认输出结构的应用,使用
-out:pdb强制输出。 - Silent 文件:使用
out:file:silent <filename>选项将输出格式更改为 Silent 文件。 - 压缩文件:使用
-out:pdb_gz生成压缩的 PDB 文件,在out:file:silent <filename>中添加.gz后缀生成压缩的 Silent 文件。
# Rosseta 输入表示
Rosetta 在处理蛋白质结构时使用两种主要表示形式:
- 全原子(full atom)
- 质心(centroid)
质心表示的必要性: 在无限时间和计算能力的理想情况下,可以对全原子进行模拟。实际上,想要采样骨架和所有的侧链原子是不现实的。计算所有原子间的相互作用复杂度约为。
质心表示: 在质心模式下,主链仍采用全原子表示,而每个侧链则简化为一个具有不同大小的伪原子。 对于蛋白质主链,这种表示方式保留了每个氨基酸的五个主链原子:氮原子(N)、α碳原子(CA)、羰基碳原子(C)、羰基氧原子(O)以及氮上的极性氢原子。此外,侧链被替换为β碳原子(CB)和一个称为 CEN (centroid atom)的伪原子,其半径和属性(如极性、电荷等)由该残基的种类决定。
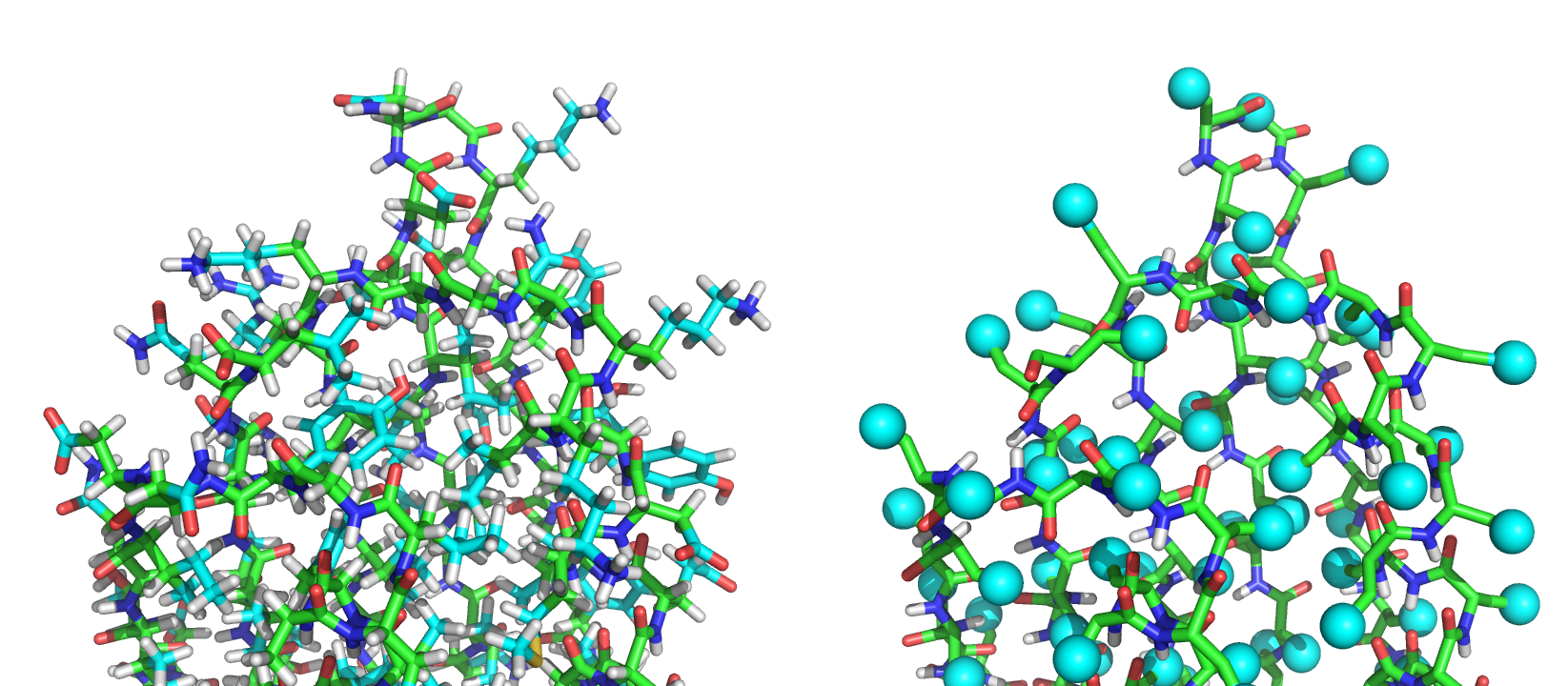
PDB 上两种模式的不同
全原子:侧链原子 CG、OD1、OD2 以及所有氢原子(如 1HB、2HB)都被明确列出。
ATOM 1 N ASP A 3 -4.524 18.589 17.199 1.00 0.00 N
ATOM 2 CA ASP A 3 -3.055 18.336 17.160 1.00 0.00 C
ATOM 3 C ASP A 3 -2.676 17.087 16.375 1.00 0.00 C
ATOM 4 O ASP A 3 -3.539 16.391 15.835 1.00 0.00 O
ATOM 5 CB ASP A 3 -2.498 18.208 18.580 1.00 0.00 C
ATOM 6 CG ASP A 3 -3.070 17.016 19.336 1.00 0.00 C
ATOM 7 OD1 ASP A 3 -3.497 16.083 18.699 1.00 0.00 O
ATOM 8 OD2 ASP A 3 -3.073 17.050 20.543 1.00 0.00 O
ATOM 9 1H ASP A 3 -4.705 19.419 17.727 1.00 0.00 H
ATOM 10 2H ASP A 3 -4.868 18.706 16.268 1.00 0.00 H
ATOM 11 3H ASP A 3 -4.985 17.814 17.630 1.00 0.00 H
ATOM 12 HA ASP A 3 -2.571 19.180 16.669 1.00 0.00 H
ATOM 13 1HB ASP A 3 -1.413 18.107 18.538 1.00 0.00 H
ATOM 14 2HB ASP A 3 -2.720 19.116 19.141 1.00 0.00 H
...
2
3
4
5
6
7
8
9
10
11
12
13
14
15
质心:侧链从 CB 开始往后的原子(如 CG、OD1、OD2 等)被省略,用一个伪原子 CEN 来近似表示整个侧链的体积和理化特性。CEN 的类型写为 X,代表它是一个伪原子,没有真实的化学对应原子。
ATOM 1 N ASP A 3 -4.524 18.589 17.199 1.00 0.00 N
ATOM 2 CA ASP A 3 -3.055 18.336 17.160 1.00 0.00 C
ATOM 3 C ASP A 3 -2.676 17.087 16.375 1.00 0.00 C
ATOM 4 O ASP A 3 -3.539 16.391 15.835 1.00 0.00 O
ATOM 5 CB ASP A 3 -2.496 18.220 18.580 1.00 0.00 C
ATOM 6 CEN ASP A 3 -2.022 18.783 19.285 1.00 0.00 **X**
ATOM 7 H ASP A 3 -5.003 18.619 18.076 1.00 0.00 H
...
2
3
4
5
6
7
8
可视化上两种模式的不同
左侧是全原子表示法,右侧质心表示法。