 Minimization Finding Deeper Energy Wells
Minimization Finding Deeper Energy Wells
# Minimizer: Finding Local Energy Minima (找到局部能量最小值)
蛋白质并不是静态结构,它们的构象会发生波动,并以一个状态集合的形式存在。 蛋白质在这些不同构象中的每一个快照都可以关联一个能量,其中一些构象具有较高的能量,而另一些则具有较低的能量。在分子建模中,通常希望找到能量函数的全局最小值(代表最低能量的构象)。全局最小值几乎是不可能完成的任务,Rosetta 来寻找一个局部最小值。 Rosetta 拥有一个核心算法,称为 minimizer,它能将结构移动到其最近的局部能量最小值。在给定起始结构的构象和能量的基础上,寻找能量函数中最近的局部最小值。
# Minimizer 最简单使用
# 设置 flags 文件
首先,需要指定如何运行最小化器。打开 minimizer_flags 文件,并分析其内容:
-s 3hon.pdb
-run:min_type lbfgs_armijo_nonmonotone
-run:min_tolerance 0.001
2
3
- 第一个参数
-s指定了输入文件,在这里是晶体结构文件3hon.pdb。 - 第二个参数
-run:min_type指定要使用的最小化算法类型,此处为lbfgs_armijo_nonmonotone。(该值是当前默认的算法类型,因此理论上可以省略该行。) - 第三个参数
-run:min_tolerance指定最小化算法的收敛容差。Rosetta 中针对函数最小化至少有两种“容差”类型:一种是“常规”容差,另一种是绝对容差。- “常规”容差是指目标函数值的相对收敛标准;例如设定容差为
0.01,意味着找到的最小函数值很可能在局部最小值的 1% 以内。 - 绝对容差则与当前函数值无关;例如设置为
0.01,则表示找到的最小值与真实局部最小值之间的差距最多为0.01REU。
- “常规”容差是指目标函数值的相对收敛标准;例如设定容差为
# 运行最小化命令
在终端窗口中,通过输入以下命令来运行最小化器可执行文件:
$> <path_to_Rosetta>/main/source/bin/minimize.default.linuxgccrelease @minimizer_flags
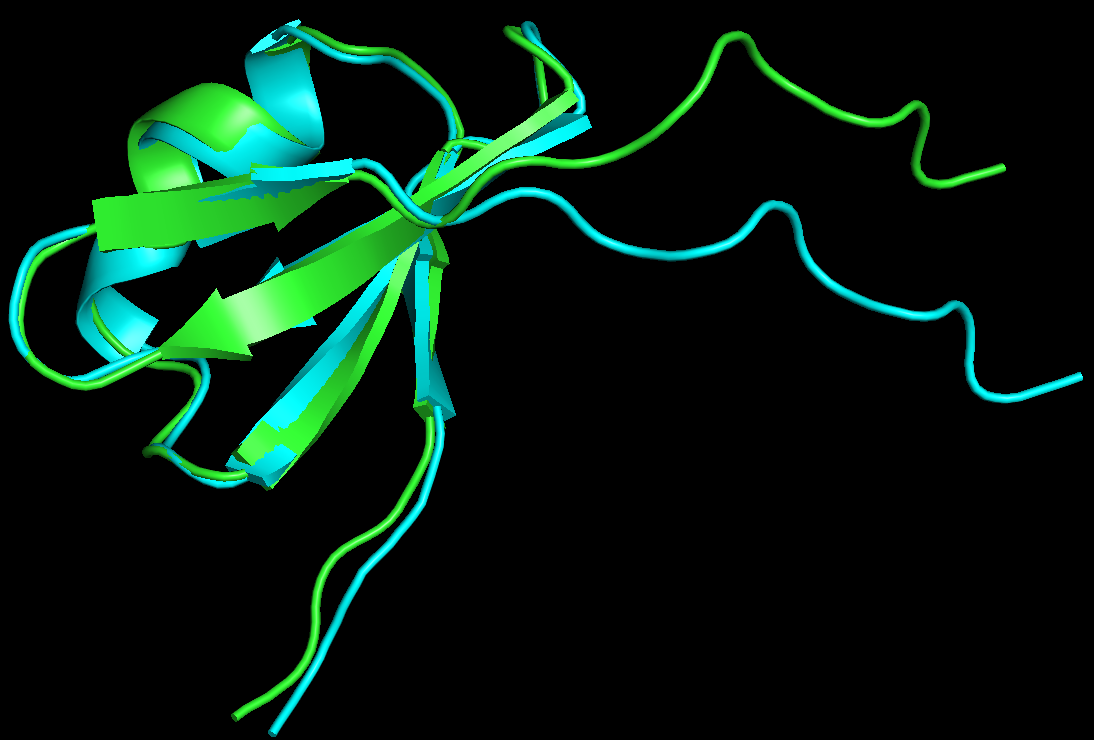
下面是最小化后的结构(绿色)已经与原始结构(青色),最后九个残基所在的 loop 区域发生了显著的构象改变。
# 带约束的最小化(进阶)
有时并不希望在起始构象中允许发生如此大的变化。为此,可以使用带约束的最小化,使输入结构在最小化过程中对某些原子的移动进行惩罚,从而限制其大幅位移。从实际操作来看,约束是在总能量函数中添加的一个额外的人工势能项(一系列惩罚偏离输入坐标的谐波势能)。
# 设置 flags 文件和约束文件
需要在 flags 文件中添加额外的选项,以告诉 Rosetta 读取约束文件,从而在最小化过程中固定部分原子的位置。此外,还需要告知 Rosetta:在使用的能量函数中加入额外的约束势能。通过在权重文件中为约束项设置非零权重值来实现这一点。
新的 flags 文件 minwithcsts_flags 如下所示:
-s 3hon.pdb
-run:min_type lbfgs_armijo_nonmonotone
-run:min_toleranace 0.001
-constraints:cst_file cstfile
-score:weights ref2015_cst
-out:suffix _minwithcsts
2
3
4
5
6
- 参数
-constraints:cst_file指定了要使用的约束文件的名称。 - 参数
-score:weights指定了能量函数中要使用的权重集合。 - 参数
-out:suffix会在输出文件名的末尾添加_minwithcsts,以避免覆盖之前的最小化结果。
# 运行最小化命令
像之前一样运行最小化可执行文件,但这次使用新的 flags 文件:
<path_to_Rosetta>/main/source/bin/minimize.default.linuxgccrelease @minwithcsts_flags
下面是最小化后的结构(绿色)已经与原始结构(紫色),最后九个残基所在的 loop 区域没有发生显著的构象改变。 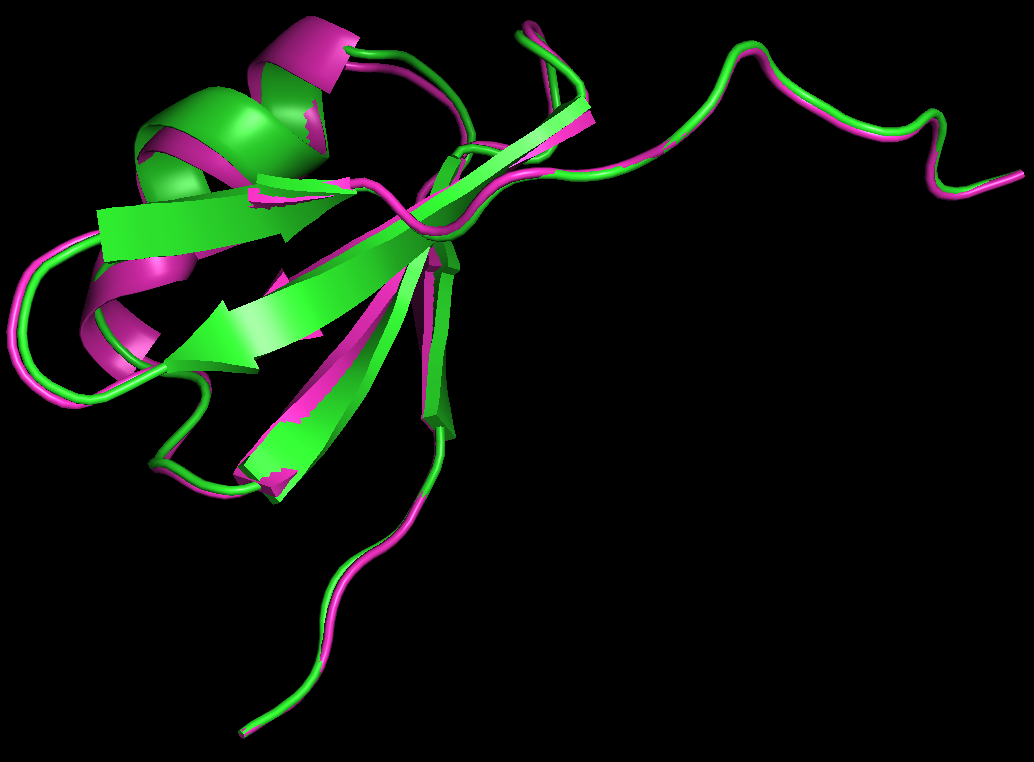
# 使用 MoveMap 进行最小化(更高阶使用)
在某些情况下,用户可能希望阻止特定残基的内部几何构型(如键角或构象角)在最小化过程中发生变化,而不仅仅是固定原子的 XYZ 坐标。为了限制主链的 phi/psi 角和/或侧链的 chi 角的变化,可以向最小化器提供一个 MoveMap,它用于指定哪些自由度可以被允许改变。
[!warning] 注意: 即使某个残基在 MoveMap 中关闭了主链和侧链的扭转角自由度,它相对于其他残基的位置仍可能发生变化,这取决于其在 FoldTree 中上游残基的运动。 FoldTree 的作用是建立结构中各个残基之间的层级关系,从而明确自由度变化时的结构响应方式。当一个残基的自由度发生变化时,这种变化会传递给它所有的子节点,但不会影响它的父节点。
[!info] MoveMap 简介 MoveMap 是 Rosetta 最小化中一个非常重要的概念。它允许用户控制哪些自由度(degrees of freedom)在最小化过程中可以改变,哪些则保持固定。 例如,在某些建模应用中,用户可能不希望移动那些高度保守的侧链;又或者在分子设计中,希望保留某些特定相互作用。某些调用 Rosetta 最小化器的协议允许使用用户自定义的 MoveMap 文件。 在最小化器的上下文中,MoveMap 让用户可以指定是否允许主链(BB)扭转角(如 α-氨基酸残基的 φ 和 ψ)以及/或者侧链(CHI)扭转角在最小化能量函数时发生变化。 此外,如果输入结构包含多个链(通过一个或多个 JUMP 或刚体变换分隔),MoveMap 还可以指定是否允许不同链之间的刚体移动。
# MoveMap 文件格式说明
MoveMap 文件中的每一行用于定义一个 jump(刚体连接)、一个残基,或一个残基范围,并指定该位置上允许的自由度(degree of freedom)。其语法格式如下:
RESIDUE <#> <BB/CHI/BBCHI/NO>
# 表示单个残基 <#>,后接一个选项(四选一)
# `BB` (Backbone):启用该残基的主链扭转角(φ 和 ψ)的优化;
# `CHI` (Sidechain):启用该残基的侧链旋转角(χ1, χ2, …)的优化;
# `BBCHI` (Backbone + Sidechain):启用该残基的主链扭转角 **和** 侧链 χ 角;
# `NO`:完全锁定该残基,不允许主链和侧链的自由度发生任何变化;
RESIDUE <#1> <#2> <BB/CHI/BBCHI/NO>
# 表示残基范围从 <#1> 到 <#2>,后接一个选项(同样四选一)
JUMP <#> <YES/NO>
# 表示 jump <#>,后接是否允许移动的选项
2
3
4
5
6
7
8
9
10
11
12
例如:
RESIDUE 28 BB # 允许第 28 个残基的主链(BB)运动
RESIDUE 32 48 BBCHI # 允许第 32 到 48 个残基的主链和侧链(CHI)运动
JUMP 1 YES # 允许 jump 1 所连接结构之间的刚体运动
2
3
也可以使用星号 * 来表示所有残基或所有 jump:
RESIDUE * CHI # 允许所有残基的侧链运动
JUMP * YES # 允许所有 jump 所连接结构之间的刚体运动
2
# 设置 movemap 文件和flags 文件,然后运行
根据自己的需求写 movemap 文件,然后在接下来的最小化操作中,需要在 flags 文件中添加一个选项,以告诉 Rosetta 读取 MoveMap 文件。新的 flags 文件 minwithmm_flags 的内容如下:
-s 3hon.pdb
-run:min_type lbfgs_armijo_nonmonotone
-run:min_toleranace 0.001
-movemap movemapfile
-out:suffix _minwithmm
2
3
4
5
其中,-movemap 参数用于指定将要应用于结构(pose)的 MoveMap 文件。
最后运行最小化命令:
<path_to_Rosetta>/main/source/bin/minimize.default.linuxgccrelease @minwithmm_flags
