Symmetry:对称
Symmetry:对称
# Symmetry:对称
许多蛋白质会组装成具有内部对称性的结构。Rosetta3 中的对称性代码开发的目的是为亚基之间的构象强制实现对称性提供一个框架。为了在 Rosetta 模拟中保持对称性,需要将三种不同类型的自由度进行对称耦合:主链、侧链和刚体。用户通常不会直接与这些底层代码交互,系统中对称性的控制和描述是通过输入的对称性定义文件来实现的。
[!note] 主链、侧链和刚体 主链自由度:蛋白质骨架的扭转角度 侧链自由度:氨基酸侧链的扭转角度 刚体自由度:亚基之间的整体平移和旋转(亚基(subunit)是指一个蛋白质复合物中单独的蛋白分子,它是组成复合物的基本单位。)
# 蛋白质中的对称性
蛋白质可以呈现多种不同类型的对称性。最常见的是环状对称性(cyclic symmetry),即围绕一个对称轴进行 n 重旋转(Cn 对称性)。另一个常见类型是二面体对称性(dihedral symmetry),它结合了一个 n 重对称轴和一个垂直的二重对称轴。更复杂的对称性还可以在立方对称群中找到。 Rosetta 能够描述所有这些类型的对称性。唯一的限制是系统中所有重复单元的化学环境必须完全相同。具备这种性质的最小重复单元就是系统的非对称单元(asymmetric unit)。非对称单元通常与一个亚基相同。
# 简单几何形状
看一个简单的对称性定义文件:
start -1,0,0 0,1,0 0,0,0
rot Rz 10
2
"start"表示第一个亚基的内部坐标系:x轴=-1,0,0,y轴=0,1,0,原点=0,0,0。z轴就是与x和y正交的向量。默认情况下,z轴是旋转轴,x轴是从亚基到(0,0,0)的连接。对称关系由rot Rz 10编码。这意味着:将360度除以10,然后绕z轴旋转这么多度数来放置下一个亚基。可以使用它将一个简单的螺旋对接到10聚体环中:
 在Rosetta中使用简单对称操作是相当有限的。例如,如果试图将三聚体和五聚体对接到某个对称阵列中,就没有单一的对称轴可以将每个亚基相互关联。
在Rosetta中使用简单对称操作是相当有限的。例如,如果试图将三聚体和五聚体对接到某个对称阵列中,就没有单一的对称轴可以将每个亚基相互关联。
# 复杂系统的基本对称性定义
# 虚拟残基的使用
为了让Rosetta移动多个蛋白质,它们必须始终以某种方式连接在一起。将内部坐标(基于扭转角)转换为3D坐标的工作原理是沿着连接"行走"并计算残基2相对于残基1的3D坐标。类似地,链B的坐标是通过从链A"行走"到链B来计算的。
在对称模式下,相关链必须相对于某个东西执行等效的移动,例如坐标系原点(0,0,0)或三个亚基之间的中点。为了实现这一点,所有亚基都必须连接到该点。实现这一目标的方法是在空间中的某个地方放置一个虚拟残基(VRT)——一个连接枢纽。然后所有亚基通过Rosetta的虚拟键(JUMP,折叠树)连接到该虚拟残基。运行带有对称性的Rosetta的技巧是将对一个跳跃(VRT --> SU_1)的所有更改应用到其他亚基(VRT --> SU_2和VRT --> SU_3)。对称性越复杂,所需要的VRT就越多。
[!note] SU_1 = 亚基1(第一个亚基),SU_2 = 亚基2(第二个亚基),SU_3 = 亚基3(第三个亚基)
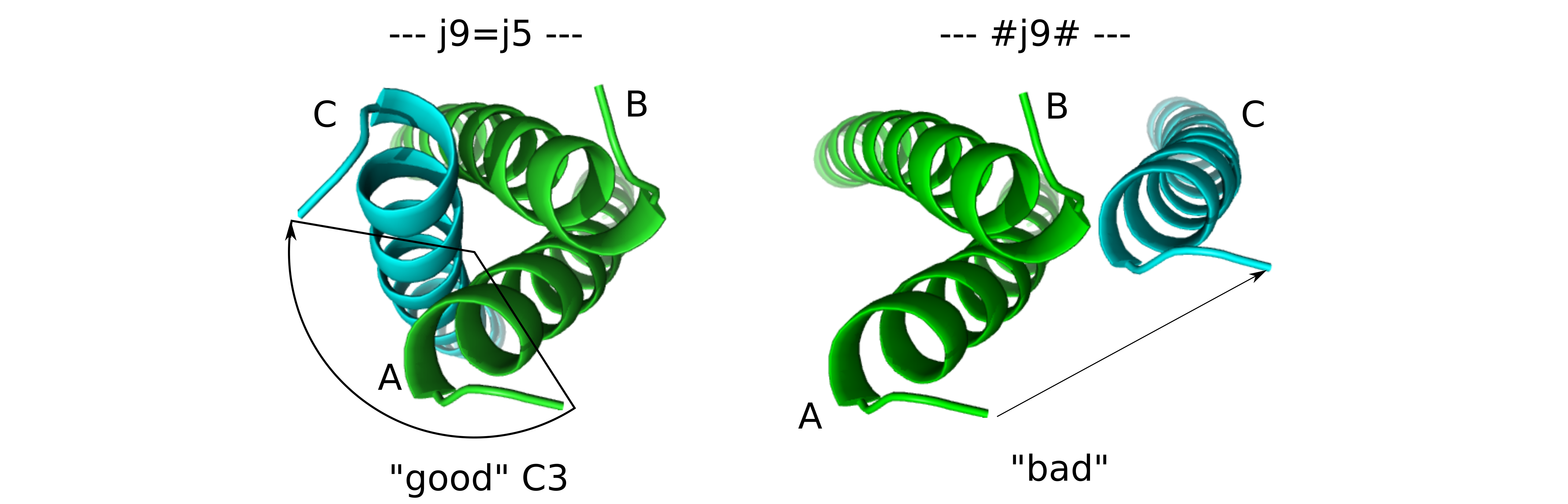
# VRTs和JUMPs - 对称折叠树
将单体2akf_INPUT.pdb转换为三聚体,忽略所输出,除了显示折叠树的部分。第一个基本上是星形的:
VRT(103)
|----=j4=---->VRT0(97)----#j5#---->VRT0_base(98)----#j1#----->18:Sub1A(1-32)
|----=j6=---->VRT1(99)----j7=5---->VRT1_base(100)----j2=1----->50:Sub2A(33-64)
\----=j8=---->VRT2(101)----j9=5---->VRT2_base(102)----j3=1----->82:Sub3A(65-96)
2
3
4
连接:
VRT0 --> (jump4) --> VRT0 --> (jump5) --> VRT0_base --> (jump1) --> 亚基1...这种连接方式对亚基2和3的连接进行了镜像。跳跃4、6和8是完全等效的。
VRT、VRT0位于相同的(中心)3D坐标处。它们对于移动链条很重要(连接跳跃的旋转和平移)。那些名为_base的是实际蛋白质亚基的连接点。
注意:
- #jumpX#标记表示可移动跳跃。
- "="符号表示对称副本。例如,跳跃7和9是跳跃5的对称副本。